Bilde 1: Sykkel-bud, av Anneli Salo (Own work) [ CC BY-SA 3.0 ], via Wikimedia Commons
Konseptet med "DNA-koden" har en lang stamtavle i genetikk. Men hva med de andre nukleinsyrer - RNA som bruker ribose i stedet for deoksyribose? Er de bare enkle transportører av biblioteket av genetisk informasjon i DNA, et ydmykt sykkel-bud i cellen? Eller har de sin egen kode? Forrige måned publiserte Nature en teknologi artikkel av Kelly Rae Chi med en spennende tittel, "RNA-koden kommer i fokus."
 Chi begynner med m6am RNA modifikasjon vi først nevnte i januar , men slutter ikke der. Modifikasjoner av RNA-baser begynner å dukke opp over alt, og deres funksjoner er bare i begynnelsen av å bli forstått. En følelse for betydningen av disse nye funnene kan fås ved å følge pengene: I de siste årene har He's gruppe oppdaget bevis som tyder på at RNA modifikasjoner gir en måte å regulere transkripsjoner involvert i brede cellulære roller, for eksempel slå på celle-differensieringsprogram. Forskere trenger bedre teknologi for å utforske disse linkene; og i oktober 2016 det amerikanske National Institutes of Health tildelt 'He og Pan' en 5-årig, US $ 10,6 millioner bevilgning for å etablere et senter til å utvikle metoder for å identifisere og kartlegge RNA modifikasjoner.
Chi begynner med m6am RNA modifikasjon vi først nevnte i januar , men slutter ikke der. Modifikasjoner av RNA-baser begynner å dukke opp over alt, og deres funksjoner er bare i begynnelsen av å bli forstått. En følelse for betydningen av disse nye funnene kan fås ved å følge pengene: I de siste årene har He's gruppe oppdaget bevis som tyder på at RNA modifikasjoner gir en måte å regulere transkripsjoner involvert i brede cellulære roller, for eksempel slå på celle-differensieringsprogram. Forskere trenger bedre teknologi for å utforske disse linkene; og i oktober 2016 det amerikanske National Institutes of Health tildelt 'He og Pan' en 5-årig, US $ 10,6 millioner bevilgning for å etablere et senter til å utvikle metoder for å identifisere og kartlegge RNA modifikasjoner.
Bilde 2. Ulike typer RNA
2. mars (2017) utstedte Japanske 'Riken lab' en nyhet som sier, "Forbedret gen-uttrykk atlas viser at mange menneskelige lange, ikke-kodende RNA faktisk kan være funksjonelle." Riken's FANTOM Consortium bygger et kart over menneskelige ikke-(protein)-kodende RNA. De nyeste funnene får en til å tenke på overraskelser med DNA i henhold ENCODE , men denne gangen med RNA i henhold til FANTOM:
Atlas, som inneholder 27 919 lange ikke-kodende RNA, oppsummerer for første gang deres uttrykksmønstre på tvers av humane celletyper og vev. Ved å krysse dette atlaset med genomiske og genetiske data, tyder deres resultater på at 19 175 av disse RNAer kan være funksjonelle, antyder at det kan være så mange - eller til og med flere - funksjonelle ikke-(protein)-kodende RNA enn de ca. 20 000 protein-kodende genene i det humane genom.
Atlaset, utgitt av Nature 9. mars, ekspanderer inn i RNA-sfæren fra funn i ENCODE og GENCODE databaser. Som med ENCODE, katalogiserer forskere så langt uttrykks-profiler uten nødvendigvis å forstå selve funksjonene. Formodentlig har cellene grunner for å uttrykke disse lange ikke-kodende RNA (lncRNAs). Jakten på de faktiske funksjoner er klar til å bære frukter, som det gjorde med ENCODE.
På samme dag (09.03), publiserte Nature en artikkel [finding] "More uses for genomic junk." Karen Adelman og Emily Egan påpeker at tidligere studier kan ha gått glipp funksjonene til "junk DNA" ved å overse nøkkelen:
I tillegg til protein-kodende messenger RNA, produserer våre celler et mangfold av forskjellige ikke-kodende RNA-molekyler. Mange av disse er generert fra sekvenser som er fjernt fra gener, og inkluderer regulatoriske DNA-sekvenser som kalles forsterkere. Transkripsjonsfaktorer bundet ved forsterkere er tenkt å regulere genuttrykk ved å loope mot gener i 3D-rom. De potensielle funksjoner av ikke-kodende forsterker RNA (eRNAs) i denne prosessen har vært ivrig debattert, men det har vært en tendens til å avskrive dem som uhell, transkriberte biprodukter av forsterker-geners interaksjoner. Tross alt, hvordan kunne korte, ustabile, heterogene RNA ha en rolle i genregulering? Ved å skrive i cellen, avslører Bose et al. at disse eRNAs kan faktisk være funksjonelle, når produsert i nærhet av enzymet CBP.
 Og hva gjøre CBP-enzymet?
Og hva gjøre CBP-enzymet?
En transkripsjons co-aktivator er acetyltransferase enzymet CBP, som sammen med sin nære slektning p300, knytter seg til DNA i forsterker regioner, hvor det legger acetylgrupper til histoner og transkripsjons-faktorer. Denne acetyleringen fremmer rekruttering av tallrike ko-transkripsjons aktivatorer og kromatin-remodellerende proteiner som har acetyl-bindende regioner, sammen med RNA-syntese av enzymet polymerase II (Pol II).
Med andre ord, trenger CBP (et protein-enzym) og forsterker RNA å være sammen for å arbeide. Implikasjonen er klart; langt fra å være utilsiktet biprodukter, er eRNAs funksjonelle. De er involvert i å lage gener tilgjengelige for oversettelses maskineriet, og regulerer deres uttrykk. Transkripsjon, lenge antatt å være motoren, er bare en del av en mye mer kompleks fabrikk.
En modell dukker opp hvor transkripsjon i seg selv er et tidlig trinn i forsterker-aktivering. Pol II er rekruttert av transkripsjonsfaktorer og opprettholder åpning av kromatin. Når enzymet begynner å transkribere, stimulerer den gryende eRNA den produserer co-aktivator proteiner som CBP i regionen på en sekvens- og stabilitets-uavhengig måte. Virksomheten til disse proteinene fremmer rekruttering av flere transkripsjonsfaktorer, Pol II og kromatin-remodeling proteiner, som muliggjør full forsterker-aktivering. I tillegg kan Pol II selv tjene som et redskap for å tiltrekke kromatin modifiserende enzymer som sprer mer molekylære merker assosiert med kromatin aktivering over transkribert region. På denne måte kan transkripsjonen av forsterkere generere en positiv tilbakekoplingssløyfe som stabiliserer både forsterker aktiviteten og gen-uttrykks-profiler.
 Totalt sett endrer denne studien fundamentalt debatten rundt eRNA funksjoner, ved å vise at disse RNA kan ha store, steds-spesifikke roller i forsterker aktivitet som ikke krever en bestemt RNA-sekvens kontekst eller overflod. Videre, ved å tilveiebringe et sterkt bevis på at CBP samvirker med eRNAs som blir transkribert, fremhever denne studien verdien av å undersøke begynnende RNAs for å forstå forsterker aktivitet.
Totalt sett endrer denne studien fundamentalt debatten rundt eRNA funksjoner, ved å vise at disse RNA kan ha store, steds-spesifikke roller i forsterker aktivitet som ikke krever en bestemt RNA-sekvens kontekst eller overflod. Videre, ved å tilveiebringe et sterkt bevis på at CBP samvirker med eRNAs som blir transkribert, fremhever denne studien verdien av å undersøke begynnende RNAs for å forstå forsterker aktivitet.
Når en snakker om 3D-rom, forskere ved Max Delbrück senter for molekylærmedisin har (MDC) har produsert et 3D-kart av genomet, som understreker det komplekse samarbeidet til DNA, RNA og proteiner:
Celler står overfor en vanskelig oppgave. De må pent pakke en flere meter lang tråd av genetisk materiale inn i en kjerne som måler bare fem mikrometer over. Denne brettekunsten skaper romlige interaksjoner mellom gener og deres brytere, noe som kan påvirke menneskers helse og sykdom. Nå har et internasjonalt team av forskere utviklet en kraftig ny teknikk som "kartlegger" denne tredimensjonale geografien til hele genomet. Deres artikkel er publisert i Nature.
Artikkelen forklarer ‘Genome Architecture Mapping’ (GAM) teknikken de skapte og hvordan den belyser samspillet mellom gener og deres forsterkere.
GAM avslører også en overflod av tre-veis kontakter over hele genomet, særlig mellom regioner som er sterkt transkriberte eller inneholder super-forsterkere, som gir en grad av innsikt i genom arkitektur som, på grunn av tekniske begrensninger i dagens teknologi, tidligere har vært uoppnåelige. Videre fremhever GAM en rolle for gen-uttrykte spesifikke kontakter i organisering av genomet i pattedyr- kjerner.
Bilde 4. Fra gen til 3-dim. protein
Er ikke det en verdig funksjon? Å holde genomet organisert er ikke en rolle som “genetisk søppel" er sannsynlig å lykkes i.
Et annet holdepunkt for å fungere i RNA kommer fra et funn annonsert av Science Daily , "Startkodonene i DNA kan være mer tallrike enn tidligere antatt." Da DNA må oversettes til messenger RNA (mRNA), ble det antatt at et 'startkodon 'identifisert starten av genet, og at det var bare syv av disse i den genetiske koden. Men ingen hadde noensinne sjekket, sier denne artikkelen. Forskere fra ‘National Institute of Standards and Technology’ fant til sin overraskelse, at det er "minst 47 mulige startkodoner, som hver kan instruere en celle til å starte proteinsyntesen." Ja, "det kan faktisk være at alle kodoner kan være startkodoner". Mulighetene dette åpner for ekspandering av kompleksiteten av RNA-transkripter kan bare tenkes på dette punktet.
 Vi skal avslutte med et eksempel på revolusjonen i RNA funksjoner. Forskere ved ‘Indiana University’ og kolleger fant et eksempel på "Hybrid inkompatibilitet forårsaket av en epi-allele." Den åpne tilgang studie, publisert i PNAS, "viser et tilfelle av epigenetisk gen-stilning snarere enn pseudogen dannelse ved mutasjon" i laboratorie fabrikken Arabidopsis. Her er en sak der RNA-halen ser ut til å logre med DNA-hunden:
Vi skal avslutte med et eksempel på revolusjonen i RNA funksjoner. Forskere ved ‘Indiana University’ og kolleger fant et eksempel på "Hybrid inkompatibilitet forårsaket av en epi-allele." Den åpne tilgang studie, publisert i PNAS, "viser et tilfelle av epigenetisk gen-stilning snarere enn pseudogen dannelse ved mutasjon" i laboratorie fabrikken Arabidopsis. Her er en sak der RNA-halen ser ut til å logre med DNA-hunden:
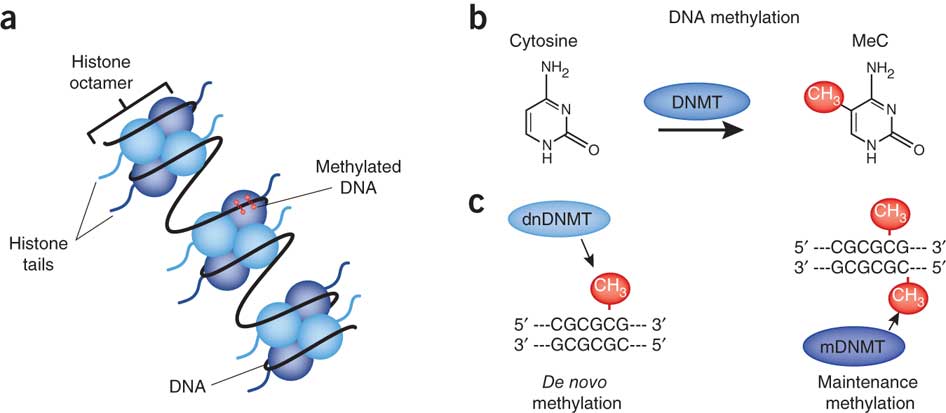
Multikopierte transgener blir ofte denaturert og brakt til taushet, særlig når de settes inn i genomet som inverterte repetisjoner som kan gi opphav til dobbel-strandet RNA. Slike dobbelt-trådet RNA kan kuttes i små interfererende RNA (siRNAs) som styrer cytosin-metylering av homologe DNA-sekvenser, en prosess som kalles Rna-(directed)rettet Dna Metylering (RdDM) .... Denne interessante case studien har vist at naturlig forekommende RdDM, som involverer en ny paralog (dupliserings begivenhet) som inaktiverer forfedrenes paralog kan på den andre siden, være en årsak til inkompatibilitet i hybridisering.
Bilde 5. Eks. på Cytosin-metylering
Omgåelse av genetiske mutasjoner og naturlig utvalg, dette "tidligere ikke epigenetisk fenomen" kan bidra til å forklare tilfeller av tilsynelatende rask artsdannelse fra en ikke-darwinistisk prosess. Vi skal overlate den muligheten til andre å utforske.
Kort sagt, har RNA gått gradene fra lærling til mester. De tallrike RNA-transkripter som flyter rundt i kjernen, en gang antatt å være genetisk "støy", kan faktisk være utførelsen, lik virtuoser et orkester bruker for å bringe statiske noter skrevet i DNA, til liv. Dette enorme skiftet i tenkning ser ut til å være dypt problematisk for neo-darwinismen. Det høres ut som en symfoni av intelligent design.
Oversatt av Asbjørn E. Lund
(Bildene sto ikke i opprinnelig artikkel, evt. untatt det 1., og er satt inn av undertegnede, se lenke i Bilde-nr)


 Chi begynner med m6am RNA modifikasjon vi først nevnte i januar , men slutter ikke der. Modifikasjoner av RNA-baser begynner å dukke opp over alt, og deres funksjoner er bare i begynnelsen av å bli forstått. En følelse for betydningen av disse nye funnene kan fås ved å følge pengene: I de siste årene har He's gruppe oppdaget bevis som tyder på at RNA modifikasjoner gir en måte å regulere transkripsjoner involvert i brede cellulære roller, for eksempel slå på celle-differensieringsprogram. Forskere trenger bedre teknologi for å utforske disse linkene; og i oktober 2016 det amerikanske National Institutes of Health tildelt 'He og Pan' en 5-årig, US $ 10,6 millioner bevilgning for å etablere et senter til å utvikle metoder for å identifisere og kartlegge RNA modifikasjoner.
Chi begynner med m6am RNA modifikasjon vi først nevnte i januar , men slutter ikke der. Modifikasjoner av RNA-baser begynner å dukke opp over alt, og deres funksjoner er bare i begynnelsen av å bli forstått. En følelse for betydningen av disse nye funnene kan fås ved å følge pengene: I de siste årene har He's gruppe oppdaget bevis som tyder på at RNA modifikasjoner gir en måte å regulere transkripsjoner involvert i brede cellulære roller, for eksempel slå på celle-differensieringsprogram. Forskere trenger bedre teknologi for å utforske disse linkene; og i oktober 2016 det amerikanske National Institutes of Health tildelt 'He og Pan' en 5-årig, US $ 10,6 millioner bevilgning for å etablere et senter til å utvikle metoder for å identifisere og kartlegge RNA modifikasjoner.  Og hva gjøre CBP-enzymet?
Og hva gjøre CBP-enzymet? Totalt sett endrer denne studien fundamentalt debatten rundt eRNA funksjoner, ved å vise at disse RNA kan ha store, steds-spesifikke roller i forsterker aktivitet som ikke krever en bestemt RNA-sekvens kontekst eller overflod. Videre, ved å tilveiebringe et sterkt bevis på at CBP samvirker med eRNAs som blir transkribert, fremhever denne studien verdien av å undersøke begynnende RNAs for å forstå forsterker aktivitet.
Totalt sett endrer denne studien fundamentalt debatten rundt eRNA funksjoner, ved å vise at disse RNA kan ha store, steds-spesifikke roller i forsterker aktivitet som ikke krever en bestemt RNA-sekvens kontekst eller overflod. Videre, ved å tilveiebringe et sterkt bevis på at CBP samvirker med eRNAs som blir transkribert, fremhever denne studien verdien av å undersøke begynnende RNAs for å forstå forsterker aktivitet. Vi skal avslutte med et eksempel på revolusjonen i RNA funksjoner. Forskere ved ‘Indiana University’ og kolleger fant et eksempel på "Hybrid inkompatibilitet forårsaket av en epi-allele." Den åpne tilgang studie, publisert i PNAS, "viser et tilfelle av epigenetisk gen-stilning snarere enn pseudogen dannelse ved mutasjon" i laboratorie fabrikken Arabidopsis. Her er en sak der RNA-halen ser ut til å logre med DNA-hunden:
Vi skal avslutte med et eksempel på revolusjonen i RNA funksjoner. Forskere ved ‘Indiana University’ og kolleger fant et eksempel på "Hybrid inkompatibilitet forårsaket av en epi-allele." Den åpne tilgang studie, publisert i PNAS, "viser et tilfelle av epigenetisk gen-stilning snarere enn pseudogen dannelse ved mutasjon" i laboratorie fabrikken Arabidopsis. Her er en sak der RNA-halen ser ut til å logre med DNA-hunden: