Når virkelighet og teori ikke går overens
(utdrag og refleksjoner ut fra ch.10 i: 'The Epigenetics Revolution'')
Noen ganger skjer det større endringer i vitenskapelige sammenhenger. Det skjedde i fysikken, da den endret seg fra 'Steady State' modellen, med et evig univers til 'Big Bang' modellen. En kan mene ulikt om denne, -og det kan synes som en må 'finne opp' mørk materie og energi -for å få teori og virkelighet til å passe, men i vår sammenheng er det viktigste at verden har en begynnelse. Alt som har en begynnelse, må nødvendigvis ha sitt opphav utenfor seg selv (Kalam kosmologiske argument).
En tilsvarende vinkling kan anlegges på livets opprinnelse. Prebiotisk seleksjon blir en selvmotsigelse, når seleksjon forutsetter en formerende organisme. Likedan for mutasjoner, hva skulle mutere når det ikke finnes liv? Det vi skal se på her angår mer hva som skiller artene, og hvorfor de er så ulike. Fra evolusjonistisk hold (0) påstås det: "Det er nå grundig dokumentert at arvelig variasjon skapes av mutasjoner, som er tilfeldige hendelser, og at variasjonen gir grunnlag for seleksjon.." Men dette utsagnet, som har fungert som 'trosgrunnlag' i over 150 år, er bare en del av virkeligheten. Ny teknologi og betydelig økt datakraft har gitt innsyn i en verden, som man før ikke ante rekkevidden av. Det er store endringer i oppfatninger av hvordan gener uttrykkes, men hva er så det spesifikt nye?
I 1965 ble Nobel Prisen i medisin delt ut for oppdagelser angående genetisk kontroll av enzymer og virus-oppbygning. Inkludert i arbeidet var oppdagelsen av 'messenger-RNA' (mRNA), som er det relativt kortlivede molekylet som overfører informasjon fra kromosomets DNA, og fungerer som en midlertidig mal for produksjon av proteiner. Vi har i mange år visst om ulike typer av RNA i cellene våre: transport-RNA (tRNA), som er små RNA-molekyler som kan knytte seg til en spesifikk aminosyre i ene enden. tRNA frakter aminosyren tilhørende mRNA til rett plass i den voksende proteinkjeden. Dette finner sted i ribosomene, cellens fabrikk, inni cytoplasmaet. RNA fra ribosomene (rRNA) er en hovedkomponent som virker som et stillas som holder flere andre RNA og protein-molekyler i rett posisjon. Dette syntes å være eksisterende RNA-verden.
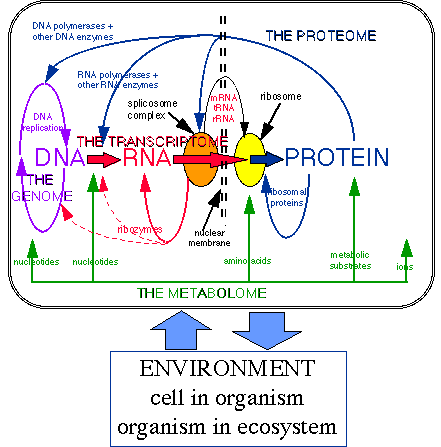 I flere tiår var DNA (underliggende kode) og proteinene (cellens fungerende molekyler), stjernene på den molekylær-biologiske catwalk. Nå er det slik at alle som arbeider i molekylærbiologi vet at proteiner er umåtelig viktige. De utfører et bredt område av funksjoner som tillater at liv opptrer. Derfor er genene som koder for proteiner også umåtelig viktige. Selv små endringer i disse protein-kodende genene kan gi ødeleggende effekter, lik mutasjoner som forårsaker blødersyke og cystisk fibrose. Men en har kanskje vært noe enøyde. At proteiner og gener som koder for det er livsviktige, medfører ikke at alt annet i genomet er uviktig. Likevel har det vært rådende oppfatning i flere tiår nå, selv om vi i mange år har visst av data, som viser at proteiner ikke utgjør helheten.
I flere tiår var DNA (underliggende kode) og proteinene (cellens fungerende molekyler), stjernene på den molekylær-biologiske catwalk. Nå er det slik at alle som arbeider i molekylærbiologi vet at proteiner er umåtelig viktige. De utfører et bredt område av funksjoner som tillater at liv opptrer. Derfor er genene som koder for proteiner også umåtelig viktige. Selv små endringer i disse protein-kodende genene kan gi ødeleggende effekter, lik mutasjoner som forårsaker blødersyke og cystisk fibrose. Men en har kanskje vært noe enøyde. At proteiner og gener som koder for det er livsviktige, medfører ikke at alt annet i genomet er uviktig. Likevel har det vært rådende oppfatning i flere tiår nå, selv om vi i mange år har visst av data, som viser at proteiner ikke utgjør helheten.
Bilde 1. Det viktige proteinet -og dets tilblivelse
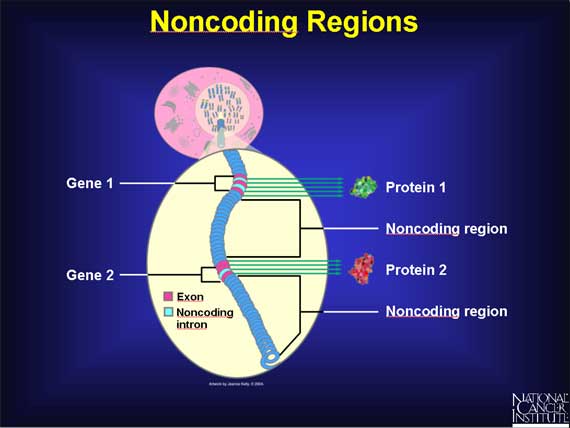
Biologer har i noen tid visst at kopien (blueprintet) av DNA blir redigert før det leveres til 'arbeidsfolkene'. Det har sammenheng med introner (in-expressed DNA). De kopieres fra DNA til mRNA, men blir spleiset ut, før meldingen blir oversatt til en protein-sekvens av ribosomet. Introner ble først identifiser i 1975 (3). Tilbake i 1970-årene sammenlignet vitenskapsfolk enkle éncellede organismer og komplekse organismer som mennesker. Mengden av DNA i dem syntes forbløffende likt, tatt i betraktning hvor ulike organismene var. Det var også da at flere gen-laboratorier viste at store deler av pattedyrs DNA inneholder sekvenser som synes å bli repetert og ikke kunne kode for protein. Dermed ble det antatt at det ikke bidro til cellens funksjoner, bare at de var med 'for turens skyld' (4,5).
 Mennesket har mellom 20 til 25 tusen gener, færre enn ei vannloppe, ris eller en banan. At mennesker skulle være mindre sofistikerte enn en banan {hva antall proteinkodende gener angår -oversetters tilføyelse}, var en av de første overraskelsene fra Human Genome Project i 2001, som satte seg fore å sekvensiere hver bokstav i menneskelig DNA. Den andre overraskelsen var at mindre enn 2% av hele genomet faktisk besto av gener, som kodet for proteiner, som er hva alt liv består av (6). Om en sammenlikner menneskets genom med det til en mikroskopisk orm, C(aenorhabditis)-elegans, så har den omtrent like mange (proteinkodende) gener som mennesket, men består bare av ca 1000 celler. Disse har vitenskapsfolk vært i stand til eksakt å identifisere, hvordan hver av dem oppstår. Selv om mye bra kan sies om C-elegans, så er den klart en mye mindre kompleks organisme enn oss mennesker. Hvorfor er vi så mye mer sofistikerte? Den opprinnelige formodning var at komplekse organismer hadde flere protein-kodende gener enn enkle organismer.
Mennesket har mellom 20 til 25 tusen gener, færre enn ei vannloppe, ris eller en banan. At mennesker skulle være mindre sofistikerte enn en banan {hva antall proteinkodende gener angår -oversetters tilføyelse}, var en av de første overraskelsene fra Human Genome Project i 2001, som satte seg fore å sekvensiere hver bokstav i menneskelig DNA. Den andre overraskelsen var at mindre enn 2% av hele genomet faktisk besto av gener, som kodet for proteiner, som er hva alt liv består av (6). Om en sammenlikner menneskets genom med det til en mikroskopisk orm, C(aenorhabditis)-elegans, så har den omtrent like mange (proteinkodende) gener som mennesket, men består bare av ca 1000 celler. Disse har vitenskapsfolk vært i stand til eksakt å identifisere, hvordan hver av dem oppstår. Selv om mye bra kan sies om C-elegans, så er den klart en mye mindre kompleks organisme enn oss mennesker. Hvorfor er vi så mye mer sofistikerte? Den opprinnelige formodning var at komplekse organismer hadde flere protein-kodende gener enn enkle organismer.
Bilde 2. Skriptet kan leses på mange måter
Slik fant man imidlertid ut at det ikke var. Mennesket har nokså likt antall gener med det ei bananflue har. Likevel vil de fleste av oss hevde at det er en betydelig forskjell på oss og en bananflue. Men hva består den i? C.elegans er en mikroskopisk orm, ca. 1 mm. lang. Selv om den har innvoller, så består den bare av ca. 1000 celler. Det var lenge antatt at økende kompleksitet skyldtes flere proteinkodende gener enn enkle organismer. Men slik skulle det ikke vise seg å være. C. elegans som er en av de best utforskede arter, viste seg å ha ca. 20.200 gener (8), altså ikke så veldig ulikt mennesket. For å se det i en sammenheng, så viser sammenligninger at jo mer komplekse organismer blir, så er det en tiltagende del av genomet som ikke koder for proteiner. Mange av proteinene i ulike høyerestående dyr er de samme, -selv om en del også er ulike. Det kan virke som slingringsmonnet og variasjonen i proteiner er lite: En liten variasjon kan føre til store defekter. Sett fra design synspunkt, er disse gjenbruk av felles design -for ulike arter. Fra evolusjonistisk hold benyttes dette for å grunngi felles avstamning.
Noe av det viktigste som ny forskning innen epigenetikk bringer oss, er alle de måter gener kan uttrykkes på. Da er det ikke lenger snakk om tilfeldige mutasjoner, som for alle praktiske forhold har vist å bringe uorden, sykdom og død. Nei, det dreier seg f.eks. om at det er mange måter å kombinere oppskriften i DNAet på. En sammenligner med script som er tilpasset de spesifikke celletyper, flere hundre i mennesket, og at RNA har en fundamentalt større betydning enn man tidliger har tillagt det. Tidligere ble RNA betraktet som midlertidige 'budbærere' (mRNA) og 'transportører' (tRNA), hvor alt dreide seg om DNA-etsproduksjon av proteiner. Bananflue har ca. 30 millioner proteinkodende basebar, mens mus bare har 26 mill. og mennesket 32 millioner. Bakterier har ca. 10% av genomet som ikke koder for proteiner, gjær ca. 30%, nevnte C. elegans har 75%, bananflue har ca. 82, 'pufferfish' har 91, mus og  menneske har ca. 98%. Vi ser at økende kompleksitet er mer i samsvar med andel av genomet som ikke koder for proteiner, enn det er med antall basepar som koder for proteiner. Det er på høy tid å rette opp i en språklig utydelighet: ikke-kodende DNA innebærer at den ikke koder FOR PROTEIN, ikke nødvendigvis at den ikke koder for noe som helst. Fravær av bevis, er ikke det samme som bevis for fravær. En må ha rett teknologi for å oppdage hva som evt. kodes. Det var ikke før ny teknologi for sporing, kombinert med enormt øket datakraft, at vi begynte å innse at noe meget interessant skjedde i de resterende 98%, den 'ikke-kodende' del av genomet.
menneske har ca. 98%. Vi ser at økende kompleksitet er mer i samsvar med andel av genomet som ikke koder for proteiner, enn det er med antall basepar som koder for proteiner. Det er på høy tid å rette opp i en språklig utydelighet: ikke-kodende DNA innebærer at den ikke koder FOR PROTEIN, ikke nødvendigvis at den ikke koder for noe som helst. Fravær av bevis, er ikke det samme som bevis for fravær. En må ha rett teknologi for å oppdage hva som evt. kodes. Det var ikke før ny teknologi for sporing, kombinert med enormt øket datakraft, at vi begynte å innse at noe meget interessant skjedde i de resterende 98%, den 'ikke-kodende' del av genomet.
Bilde 3. Dess mer komplekst, desto mer ikke-protein-kodende DNA
Av de 98% av genomet som ikke koder for protein, trodde man tidligere, i tråd med hypotesen om felles avstamning, at 90% var overlevning fra tidligere organismer, såkalt søppel-DNA. Nå er man kommet til bortimot motsatt resultat: Minst 80, kanskje nærmere 90% av DNAet, har en funksjon. Og stadig finner en nye sammenhenger og funksjoner. I korthet går det altså på at det produseres RNA, som viser seg å ha viktige funksjoner i livssyklusen, dannet av det 'ikke-kodende' DNAet. RNA som dannes, kan være både lengre sekvenser, på flere tusen baser, eller som vi senere kan se på: tusenvis av korte micro-RNA. Det er viktig å poengtere at alt som foregår i cellen, skjer i samarbeid mellom RNA, DNA og proteiner. Der er ingen solospillere, men komplekse og spesifiserte informasjonssystem, hvor alle delene er nødvendige, og det er vanskelig å forestille seg hvordan de kunne klare seg uten noen av dem.
Vi skal vise et eksempel på dette, overført til betydninger hentet fra det norske språket (Fig. 1). Det initielle mRNA inneholder alle exoner og introner. Så spleises det for å fjerne intronene. Men i løpet av denne spleisingen, kan også noen av exonene bli fjernet. Noen exoner vil bli opprettholdt i det endelige mRNA, mens andre droppes. På Figur 1 vises exonene, som koder for aminosyrere -i bokser med bokstaver. De tomme boksene tilsvarer introner. Ved første kopiering beholdes både exoner og introner, så fjerner det cellulære maskineriet noen eller alle intronene (spleising). De resulterende mRNA molekylene, kan kode for mange proteiner fra det samme genet, som vist ved de ulike ordene i diagrammet. For å ta C. elegans så kan den kanskje kode for 'MIDD' og 'DEL', mens mennesket ved spleising kan kode for samtlige oppførte 'proteiner'.
| M |
|
I |
|
D |
|
D |
|
E |
|
L |
|
H |
|
A |
|
V |
|
E |
|
T |
1. Kopiering
|
| M |
|
I |
|
D |
|
D |
|
E |
|
L |
|
H |
|
A |
|
V |
|
E |
|
T |
koder for: |
| |
|
|
|
|
|
|
|
M |
I |
D |
D |
|
|
|
|
|
|
|
|
|
+
|
| |
|
|
|
|
|
|
|
|
I |
D |
|
|
|
|
|
|
|
|
|
|
+ |
| |
|
|
|
|
|
|
|
|
D |
E |
L |
|
|
|
|
|
|
|
|
|
+ |
| |
|
|
|
|
|
|
|
|
H |
A |
V |
E |
|
|
|
|
|
|
|
|
+ |
| |
|
|
|
|
|
|
|
H |
A |
V |
E |
T |
|
|
|
|
|
|
|
|
+ |
| |
|
|
|
|
M |
I |
D |
D |
E |
L |
H |
A |
V |
E |
T |
|
|
|
|
|
Figur 1. Spleising og redigering-hvordan ett gen kan kode for mange proteiner
Med mye forbedrede metodene begynte en å innse at det faktisk var mye avskriving som pågikk i deler av genomet, som IKKE var PROTEIN-kodende. I starten avfeide en dette med at det var 'støy', helt i tråd med gjeldende 'paradigme'. En mente at selv om en kunne oppdage disse molekylene med nytt, sensitivt utstyr, så bidro de ikke biologisk sett. Nå var det imidlertid flere forhold som antydet at denne formen for transkripsjon slett ikke var tilfeldig (9). Mønstrene til disse ikke-protein-kodende RNA, var f.eks. ulike for ulike celletyper. Det fantes mange slike mønstre i hjernen, og de var ulike i ulike regioner av hjernen (10). Denne effekten er reproduserbar, når hjerne-regioner blir sammenlignet for ulike individer. Det er ikke i tråd med hva en ville forvente om denne lav-nivå avskrivningen av RNA bare var en helt tilfeldig prosess.
 Bilde 4. Ikke-PROTEIN-kodende regioner
Bilde 4. Ikke-PROTEIN-kodende regioner
Endringen i fokus foregår ved at en flytter blikket fra proteiner og gener som er protein-kodende, til den nesten hele delen av genomet som ikke er det. Noen slike ikke-PROTEIN-kodende RNA kommer fra introner (som regnes som 'inexpressed'-'ikke uttrykt' i proteiner). Andre overlapper gener, ofte skrevet av fra motsatt side av protein-kodende mRNA. Andre igjen finnes i regioner hvor det ikke er noen protein-kodende gener i det hele. For ikke-PROTEIN-kodende RNA begynner endelig å stilles jamsides proteiner, som fullt fungerende molekyler. Ulike, men likeverdige. Vi skal se på noen konkretiseringer av ikke protein-kodende RNA: Xist og Tsix, som kreves for å inaktivere X-kromosomet. Begge er lange RNA, på flere tusen kilobaser i lengde. DA Xist ble identifisert, var den bare den andre av ikke-protein-kodende RNAs. Nåværende estimat går i retning av at det finnes flere tusen slike molekyler i celler av høyere pattedyr, lengre enn de som er rapportert i mus (11). Lange ikke-protein-kodende RNA kan faktisk være flere enn protein-kodende mRNAs, hvorav Xist RNA kan være en av de viktigste (12A).
I tillegg til deaktivering, synes lange RNA også å spille en kritisk rolle i å merke gen-avtrykk. Mange merkede regioner inneholder inneholder en seksjon som koder for et langt, ikke-protein-kodende RNA, i likhet med virkningen av Xist. I mus er ett ikke-protein-kodende RNA, som kalles 'Air' uttrykt i morkaken, eksklusivt arvet fra farens kromsom (11). Air fører til at nærliggende gen (Ifg2r) stilnes, men bare på samme kromosom (12B). Således bevirker Air at Igfr2 bare uttrykkes fra det kromosomet som arves fra moren. Slikt bidro til å kaste lys over spørsmål som epigenetikere hadde lurt på: 'Hvordan plasseres histon-modifiserende enzymer, som setter eller fjerner epigenetiske markører, på spesifikke regioner i genomet?'
 Air ble lokalisert til en spesifikk region i klusteret av merkede gener, og virket som en magnet på ett epigenetisk enzym (G9a). Dette enzymet deaktiverer H3 proteiner i histonet, i denne DNA-regionen av nukleosomet. Denne histon-modifiseringen skapet en undertrykkende kromatin-omgivelse, som slår av gener. Kromatin (lenke) er i seg selv interessant, som ett kompleks av makromolyler i celler, som består av både DNA, proteiner og RNA. Hovedfunksjonene til kromatin er: i) å pakke DNA i mindre volum for å passe i cellen ii) å forsterke DNA makromolekylet for å tillate celledeling og iii) å forhindre DNA-skade iv) å kontrollere hvordan gener uttrykkes og DNA kopieres. Den primære protein-komponenten til kromatin er histoner som fortetter DNA. Det finnes bare i celler med kjerne (eukaryote). {Det ville være bemerkelsesverdig om cellen klarte seg uten kromatin, men å utvikle noe som samtidig består av både DNA, proteiner og RNA -vil forutsette at disse finnes, liksom livet som helhet består av alle tre -oversetters anmerkning.}
Air ble lokalisert til en spesifikk region i klusteret av merkede gener, og virket som en magnet på ett epigenetisk enzym (G9a). Dette enzymet deaktiverer H3 proteiner i histonet, i denne DNA-regionen av nukleosomet. Denne histon-modifiseringen skapet en undertrykkende kromatin-omgivelse, som slår av gener. Kromatin (lenke) er i seg selv interessant, som ett kompleks av makromolyler i celler, som består av både DNA, proteiner og RNA. Hovedfunksjonene til kromatin er: i) å pakke DNA i mindre volum for å passe i cellen ii) å forsterke DNA makromolekylet for å tillate celledeling og iii) å forhindre DNA-skade iv) å kontrollere hvordan gener uttrykkes og DNA kopieres. Den primære protein-komponenten til kromatin er histoner som fortetter DNA. Det finnes bare i celler med kjerne (eukaryote). {Det ville være bemerkelsesverdig om cellen klarte seg uten kromatin, men å utvikle noe som samtidig består av både DNA, proteiner og RNA -vil forutsette at disse finnes, liksom livet som helhet består av alle tre -oversetters anmerkning.}
Bilde 5. Histon spoler -fra 'DNA and Beyond'
Mønstre med histon-modifiseringer er lokalisert i ulike gener, i ulike celle-typer og leder til utsøkte, vel-regulerte gen-uttrykk. F.eks. har enzymet EZH2 som målskive ulike H3 molekyler i ulike celle-typer. F.eks. kan det metylere H3 proteiner plassert på gen A i hvite blodceller, men ikke i nevroner. Alternativt kan det metylere H3 proteiner plassert på gen B i nevroner, men ikke i hvite blodceller. Samme enzym i begge celler, blir styrt til ulikt sted. Det er økende bevis for at i det minste noe av målstyringen for epigenetiske modifikasjoner kan bli uttrykt ved interaksjoner av lange, ikke-protein-kodende RNA. F.eks. er det nylig undersøkt lange, ikke-protein-kodende RNA, som binder seg til et kompleks av proteiner (PRC2), og som igjen genererer repressive modifikasjoner på histonet. Ved forsøk på mus fant forskere at PRC2 bandt seg til tusener av ulike, ikke-protein-kodende RNA i stamceller (13). Proteinet som interagerer med det lange, ikke-protein-kodende RNA, er antageligvis EZH2. Slike lange, ikke-protein-kodende RNA kan fungere som 'agn'. De kan befinne seg bundet til spesifikke regioner i genomet, der de er produsert, og så tiltrekke enzymer som virker repressivt og slår av genuttrykk. Det skjer fordi slike enzym-komplekser, som virker repressivt inneholder proteiner lik EZH2, som er i stand til å binde seg til DNA.
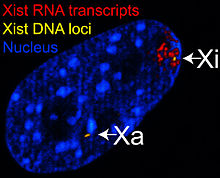 Selv om Xist er den best undersøkte av alle lange ikke-protein-kodende RNA, så er det mysterier knyttet til denne. Den brer seg utover nesten hele det inaktive X-kromosomet, men det er ennå ikke kjent hvordan. Kromosomer kveles vanligvis ikke av RNA-molekyler. Det er ingen åpenbar grunn til at Xist skulle være i stand til å binde seg slik, men vi vet det ikke har noe med gensekvensen i kromosomet. Når det har funnet feste på et kromosom, fortsetter Xist å bre seg, så sant det fantes et X-inaktiverings senter på autosomet (15). I 2010 identifiserte professor Ramin Shiekhattar ved Wistar Institute i Philadelphia over 3.000 lange, ikke-protein-kodende RNA i flere menneskelige celle-typer. De benyttet vel-etablerte metoder for å slå ut uttrykk av sine ikke-protein-kodende (ipk) RNA. Forventet resultat var annerledes enn predikert. De fant at ca. 50% av lange (ipk)RNA faktisk kunne øke gen-uttrykket for nabogener (16). Fortsetter i neste innlegg.
Selv om Xist er den best undersøkte av alle lange ikke-protein-kodende RNA, så er det mysterier knyttet til denne. Den brer seg utover nesten hele det inaktive X-kromosomet, men det er ennå ikke kjent hvordan. Kromosomer kveles vanligvis ikke av RNA-molekyler. Det er ingen åpenbar grunn til at Xist skulle være i stand til å binde seg slik, men vi vet det ikke har noe med gensekvensen i kromosomet. Når det har funnet feste på et kromosom, fortsetter Xist å bre seg, så sant det fantes et X-inaktiverings senter på autosomet (15). I 2010 identifiserte professor Ramin Shiekhattar ved Wistar Institute i Philadelphia over 3.000 lange, ikke-protein-kodende RNA i flere menneskelige celle-typer. De benyttet vel-etablerte metoder for å slå ut uttrykk av sine ikke-protein-kodende (ipk) RNA. Forventet resultat var annerledes enn predikert. De fant at ca. 50% av lange (ipk)RNA faktisk kunne øke gen-uttrykket for nabogener (16). Fortsetter i neste innlegg.
Bilde 6. Xist RNA-transkript
Referanser:
1. Fra Scientific Autobiography and Other Papers (1950)
2. Mulder et al (1975), Cold Spring Harb Symp Quant Biol. 39: 397-400
3. Ohno (1972) Brookhaven Symposium in Biology 23: 366-370
4. Orgel og Crick (1980), Nature 284: 604-607
5. Doolittle og Sapienza, (1980), Nature 284: 601-603
8. http://wiki.wormbase.org/index.php/WS205
9. Quersh et al. (2010), Brain Research 1338: 20-35
10. Clark og Mattick (2011), Seminar in Cell and Development Biology
11. Carnici et al. (2005), Science 309: 1559-1563
12A. Erwin og Lee, (2008)
12B. Nagano et al. (2005), Science 322: 1717-1720
13. Zhao et al (2010), Molecular Cell 40: 939-953
16. Ørom et al. (2005)m Cekk 143; 46.58
Stoffutvalg og bilder ved Asbjørn E. Lund
 I flere tiår var DNA (underliggende kode) og proteinene (cellens fungerende molekyler), stjernene på den molekylær-biologiske catwalk. Nå er det slik at alle som arbeider i molekylærbiologi vet at proteiner er umåtelig viktige. De utfører et bredt område av funksjoner som tillater at liv opptrer. Derfor er genene som koder for proteiner også umåtelig viktige. Selv små endringer i disse protein-kodende genene kan gi ødeleggende effekter, lik mutasjoner som forårsaker blødersyke og cystisk fibrose. Men en har kanskje vært noe enøyde. At proteiner og gener som koder for det er livsviktige, medfører ikke at alt annet i genomet er uviktig. Likevel har det vært rådende oppfatning i flere tiår nå, selv om vi i mange år har visst av data, som viser at proteiner ikke utgjør helheten.
I flere tiår var DNA (underliggende kode) og proteinene (cellens fungerende molekyler), stjernene på den molekylær-biologiske catwalk. Nå er det slik at alle som arbeider i molekylærbiologi vet at proteiner er umåtelig viktige. De utfører et bredt område av funksjoner som tillater at liv opptrer. Derfor er genene som koder for proteiner også umåtelig viktige. Selv små endringer i disse protein-kodende genene kan gi ødeleggende effekter, lik mutasjoner som forårsaker blødersyke og cystisk fibrose. Men en har kanskje vært noe enøyde. At proteiner og gener som koder for det er livsviktige, medfører ikke at alt annet i genomet er uviktig. Likevel har det vært rådende oppfatning i flere tiår nå, selv om vi i mange år har visst av data, som viser at proteiner ikke utgjør helheten. Mennesket har mellom 20 til 25 tusen gener, færre enn ei vannloppe, ris eller en banan. At mennesker skulle være mindre sofistikerte enn en banan {hva antall proteinkodende gener angår -oversetters tilføyelse}, var en av de første overraskelsene fra Human Genome Project i 2001, som satte seg fore å sekvensiere hver bokstav i menneskelig DNA. Den andre overraskelsen var at mindre enn 2% av hele genomet faktisk besto av gener, som kodet for proteiner, som er hva alt liv består av (6). Om en sammenlikner menneskets genom med det til en mikroskopisk orm, C(aenorhabditis)-elegans, så har den omtrent like mange (proteinkodende) gener som mennesket, men består bare av ca 1000 celler. Disse har vitenskapsfolk vært i stand til eksakt å identifisere, hvordan hver av dem oppstår. Selv om mye bra kan sies om C-elegans, så er den klart en mye mindre kompleks organisme enn oss mennesker. Hvorfor er vi så mye mer sofistikerte? Den opprinnelige formodning var at komplekse organismer hadde flere protein-kodende gener enn enkle organismer.
Mennesket har mellom 20 til 25 tusen gener, færre enn ei vannloppe, ris eller en banan. At mennesker skulle være mindre sofistikerte enn en banan {hva antall proteinkodende gener angår -oversetters tilføyelse}, var en av de første overraskelsene fra Human Genome Project i 2001, som satte seg fore å sekvensiere hver bokstav i menneskelig DNA. Den andre overraskelsen var at mindre enn 2% av hele genomet faktisk besto av gener, som kodet for proteiner, som er hva alt liv består av (6). Om en sammenlikner menneskets genom med det til en mikroskopisk orm, C(aenorhabditis)-elegans, så har den omtrent like mange (proteinkodende) gener som mennesket, men består bare av ca 1000 celler. Disse har vitenskapsfolk vært i stand til eksakt å identifisere, hvordan hver av dem oppstår. Selv om mye bra kan sies om C-elegans, så er den klart en mye mindre kompleks organisme enn oss mennesker. Hvorfor er vi så mye mer sofistikerte? Den opprinnelige formodning var at komplekse organismer hadde flere protein-kodende gener enn enkle organismer.  menneske har ca. 98%. Vi ser at økende kompleksitet er mer i samsvar med andel av genomet som ikke koder for proteiner, enn det er med antall basepar som koder for proteiner. Det er på høy tid å rette opp i en språklig utydelighet: ikke-kodende DNA innebærer at den ikke koder FOR PROTEIN, ikke nødvendigvis at den ikke koder for noe som helst. Fravær av bevis, er ikke det samme som bevis for fravær. En må ha rett teknologi for å oppdage hva som evt. kodes. Det var ikke før ny teknologi for sporing, kombinert med enormt øket datakraft, at vi begynte å innse at noe meget interessant skjedde i de resterende 98%, den 'ikke-kodende' del av genomet.
menneske har ca. 98%. Vi ser at økende kompleksitet er mer i samsvar med andel av genomet som ikke koder for proteiner, enn det er med antall basepar som koder for proteiner. Det er på høy tid å rette opp i en språklig utydelighet: ikke-kodende DNA innebærer at den ikke koder FOR PROTEIN, ikke nødvendigvis at den ikke koder for noe som helst. Fravær av bevis, er ikke det samme som bevis for fravær. En må ha rett teknologi for å oppdage hva som evt. kodes. Det var ikke før ny teknologi for sporing, kombinert med enormt øket datakraft, at vi begynte å innse at noe meget interessant skjedde i de resterende 98%, den 'ikke-kodende' del av genomet. Bilde 4. Ikke-PROTEIN-kodende regioner
Bilde 4. Ikke-PROTEIN-kodende regioner
 Air ble lokalisert til en spesifikk region i klusteret av merkede gener, og virket som en magnet på ett epigenetisk enzym (G9a). Dette enzymet deaktiverer H3 proteiner i histonet, i denne DNA-regionen av nukleosomet. Denne histon-modifiseringen skapet en undertrykkende kromatin-omgivelse, som slår av gener. Kromatin (lenke) er i seg selv interessant, som ett kompleks av makromolyler i celler, som består av både DNA, proteiner og RNA. Hovedfunksjonene til kromatin er: i) å pakke DNA i mindre volum for å passe i cellen ii) å forsterke DNA makromolekylet for å tillate celledeling og iii) å forhindre DNA-skade iv) å kontrollere hvordan gener uttrykkes og DNA kopieres. Den primære protein-komponenten til kromatin er histoner som fortetter DNA. Det finnes bare i celler med kjerne (eukaryote). {Det ville være bemerkelsesverdig om cellen klarte seg uten kromatin, men å utvikle noe som samtidig består av både DNA, proteiner og RNA -vil forutsette at disse finnes, liksom livet som helhet består av alle tre -oversetters anmerkning.}
Air ble lokalisert til en spesifikk region i klusteret av merkede gener, og virket som en magnet på ett epigenetisk enzym (G9a). Dette enzymet deaktiverer H3 proteiner i histonet, i denne DNA-regionen av nukleosomet. Denne histon-modifiseringen skapet en undertrykkende kromatin-omgivelse, som slår av gener. Kromatin (lenke) er i seg selv interessant, som ett kompleks av makromolyler i celler, som består av både DNA, proteiner og RNA. Hovedfunksjonene til kromatin er: i) å pakke DNA i mindre volum for å passe i cellen ii) å forsterke DNA makromolekylet for å tillate celledeling og iii) å forhindre DNA-skade iv) å kontrollere hvordan gener uttrykkes og DNA kopieres. Den primære protein-komponenten til kromatin er histoner som fortetter DNA. Det finnes bare i celler med kjerne (eukaryote). {Det ville være bemerkelsesverdig om cellen klarte seg uten kromatin, men å utvikle noe som samtidig består av både DNA, proteiner og RNA -vil forutsette at disse finnes, liksom livet som helhet består av alle tre -oversetters anmerkning.} Selv om Xist er den best undersøkte av alle lange ikke-protein-kodende RNA, så er det mysterier knyttet til denne. Den brer seg utover nesten hele det inaktive X-kromosomet, men det er ennå ikke kjent hvordan. Kromosomer kveles vanligvis ikke av RNA-molekyler. Det er ingen åpenbar grunn til at Xist skulle være i stand til å binde seg slik, men vi vet det ikke har noe med gensekvensen i kromosomet. Når det har funnet feste på et kromosom, fortsetter Xist å bre seg, så sant det fantes et X-inaktiverings senter på autosomet (15). I 2010 identifiserte professor Ramin Shiekhattar ved Wistar Institute i Philadelphia over 3.000 lange, ikke-protein-kodende RNA i flere menneskelige celle-typer. De benyttet vel-etablerte metoder for å slå ut uttrykk av sine ikke-protein-kodende (ipk) RNA. Forventet resultat var annerledes enn predikert. De fant at ca. 50% av lange (ipk)RNA faktisk kunne øke gen-uttrykket for nabogener (16). Fortsetter i neste innlegg.
Selv om Xist er den best undersøkte av alle lange ikke-protein-kodende RNA, så er det mysterier knyttet til denne. Den brer seg utover nesten hele det inaktive X-kromosomet, men det er ennå ikke kjent hvordan. Kromosomer kveles vanligvis ikke av RNA-molekyler. Det er ingen åpenbar grunn til at Xist skulle være i stand til å binde seg slik, men vi vet det ikke har noe med gensekvensen i kromosomet. Når det har funnet feste på et kromosom, fortsetter Xist å bre seg, så sant det fantes et X-inaktiverings senter på autosomet (15). I 2010 identifiserte professor Ramin Shiekhattar ved Wistar Institute i Philadelphia over 3.000 lange, ikke-protein-kodende RNA i flere menneskelige celle-typer. De benyttet vel-etablerte metoder for å slå ut uttrykk av sine ikke-protein-kodende (ipk) RNA. Forventet resultat var annerledes enn predikert. De fant at ca. 50% av lange (ipk)RNA faktisk kunne øke gen-uttrykket for nabogener (16). Fortsetter i neste innlegg.